Forschungsschwerpunkte
Wir beschäftigen uns insbesondere mit:
- Entwicklung von robusten, generalisierbaren Neuronalen Netzen (CNNs, Deep Learning)
- Daten-/Annotation-effiziente Modelle basierend auf Semi-/Self-supervised Learning
- Outlier-Detektion und Imputation von unvollständige Datensätzen
- Rekonstruktion von Bild- und Videodaten, eg. mit Hilfe von Super-Resolution
- Segmentierungsprobleme, insbesondere MRI Brain Segmentation
- Quantifizierung von Unsicherheiten von Klassifizierungsvorhersagen
- Entwicklung von interpretierbaren Features zur Verbesserung der Anwender-/Patientenkommunikation
- Evaluierung von Algorithmus Performance und Quantifizierung von Data-biases
- Translation von Forschungsergebnissen in industrielle oder medizinische Kontexte
- Quantifizierung von menschlicher Anatomie anhand von Bilddaten (MRI, X-Ray, CT) im Kontext von Erkrankungen wie Demenz, Tumoren und Traumata.
Ausgewählte Forschungsarbeiten
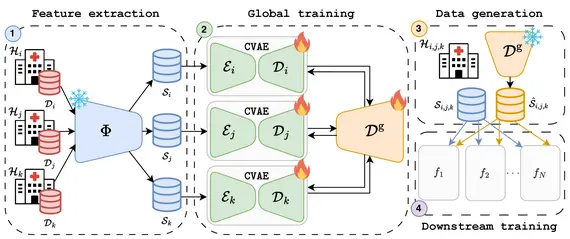
F. Di Salvo, M. Nguyen, C. Ledig, "Embedding-Based Federated Data Sharing via Differentially Private Conditional VAEs", MICCAI 2025
Deep Learning (DL) hat die medizinische Bildgebung revolutioniert. Doch seine Einführung wird durch Datenknappheit und Datenschutzbestimmungen eingeschränkt, wodurch der Zugang zu vielfältigen Datensätzen begrenzt ist. Federated Learning (FL) ermöglicht zwar ein dezentrales Training, leidet jedoch unter hohen Kommunikationskosten und ist oft auf eine einzige nachgelagerte Aufgabe beschränkt, was die Flexibilität einschränkt. Wir schlagen eine Methode zum Datenaustausch über differentiell private (DP) generative Modelle vor. Mithilfe von Foundation-Modellen extrahieren wir kompakte, informative Einbettungen, wodurch Redundanzen reduziert und der Rechenaufwand gesenkt werden. Kunden trainieren gemeinsam einen Differentially Private Conditional Variational Autoencoder (DP-CVAE), um eine globale, datenschutzbewusste Datenverteilung zu modellieren, die verschiedene nachgelagerte Aufgaben unterstützt. Unser Ansatz, der für mehrere Feature-Extraktoren validiert wurde, verbessert den Datenschutz, die Skalierbarkeit und die Effizienz. Er übertrifft dabei herkömmliche FL-Klassifikatoren, während gleichzeitig die differentielle Privatsphäre gewährleistet ist. Darüber hinaus erzeugt der DP-CVAE-Embedder mit höherer Genauigkeit als der DP-CGAN-Embedder und benötigt dabei fünfmal weniger Parameter.
Autoren: Francesco Di Salvo*, My Nguyen*, Christian Ledig
* gemeinsame Urheberschaft
[Preprint], [Bibtex](261.0 B)
F. Di Salvo, S. Doerrich, I. Rieger, C. Ledig, "An Embedding is Worth a Thousand Noisy Labels," TMLR 2025
Die Leistung tiefer neuronaler Netze skaliert mit der Größe des Datensatzes und der Qualität der Labels. Daher ist eine effiziente Reduzierung von Datenannotationen geringer Qualität für den Aufbau robuster und kosteneffizienter Systeme von entscheidender Bedeutung. Aktuelle Ansätze zur Reduktion von Label-Rauschen sind aufgrund ihrer hohen rechnerischen Komplexität und spezifischen Anwendungsanforderungen stark begrenzt. In dieser Arbeit präsentieren wir WANN, einen gewichteten adaptiven Nearest-Neighbor-Ansatz, der selbstüberwachte Merkmalsdarstellungen verwendet, die aus Basis-Modellen abgeleitet werden. Zur Steuerung des gewichteten Abstimmungsschemas führen wir einen Zuverlässigkeitswert ein. Dieser misst die Wahrscheinlichkeit, dass ein Datenlabel korrekt ist. WANN übertrifft Referenzmethoden – einschließlich einer linearen Schicht, die mit robusten Verlustfunktionen trainiert wurde – bei verschiedenen Datensätzen unterschiedlicher Größe sowie unter verschiedenen Arten und Schweregraden von Rauschen. WANN zeigt auch eine überlegene Generalisierung bei unausgewogenen Daten im Vergleich zu adaptiven neuronalen Netzen (ANN) und festen k-NNs. Darüber hinaus verbessert das vorgeschlagene Gewichtungsschema die überwachte Dimensionsreduktion bei verrauschten Labels. Dies führt zu einer deutlichen Steigerung der Klassifizierungsleistung bei 10-mal und 100-mal kleineren Bild-Embeddings, wodurch sich Latenz und Speicheranforderungen minimieren lassen. Unser Ansatz, der Effizienz und Erklärbarkeit in den Vordergrund stellt, erweist sich somit als einfache und robuste Lösung zur Überwindung der inhärenten Einschränkungen des Trainings tiefer neuronaler Netze.
Autoren: Francesco Di Salvo, Sebastian Doerrich, Ines Rieger, Christian Ledig
[Preprint], [Publication], [Code], [Bibtex](309.0 B)
S. Doerrich, F. Di Salvo, J. Brockmann, C. Ledig, “Rethinking model prototyping through the MedMNIST+ dataset collection”, Scientific Reports, 15, 7669, 2025
Die Integration von auf Deep Learning basierenden Systemen in die klinische Praxis wird häufig durch Herausforderungen behindert, die in begrenzten und heterogenen medizinischen Datensätzen begründet sind. Darüber hinaus wird in diesem Bereich zunehmend marginalen Leistungssteigerungen bei einigen wenigen, eng gefassten Benchmarks Vorrang vor der klinischen Anwendbarkeit eingeräumt, wodurch sinnvolle algorithmische Fortschritte verlangsamt werden. Dieser Trend führt häufig zu einer übermäßigen Feinabstimmung bestehender Methoden an ausgewählten Datensätzen, anstatt klinisch relevante Innovationen zu fördern. Als Reaktion darauf wird in dieser Arbeit ein umfassender Benchmark für die MedMNIST+-Datensatzsammlung eingeführt, der die Bewertungslandschaft über mehrere Bildgebungsmodalitäten, anatomische Regionen, Klassifizierungsaufgaben und Stichprobengrößen hinweg diversifizieren soll. Wir bewerten systematisch häufig verwendete Convolutional Neural Networks (CNNs) und Vision Transformer (ViT)-Architekturen in verschiedenen medizinischen Datensätzen, Trainingsmethoden und Eingabeauflösungen, um bestehende Annahmen über die Effektivität und Entwicklung von Modellen zu validieren und zu verfeinern. Unsere Ergebnisse deuten darauf hin, dass rechnerisch effiziente Trainingsverfahren und moderne Foundation-modelle praktikable Alternativen zum kostspieligen End-to-End-Training bieten. Darüber hinaus stellen wir fest, dass höhere Bildauflösungen die Leistung ab einem bestimmten Schwellenwert nicht durchgängig verbessern. Dies unterstreicht die potenziellen Vorteile der Verwendung niedrigerer Auflösungen, insbesondere in der Prototyping-Phase, um den Rechenaufwand zu verringern, ohne die Genauigkeit zu beeinträchtigen. Insbesondere bestätigt unsere Analyse die Wettbewerbsfähigkeit von CNNs im Vergleich zu ViTs und unterstreicht, wie wichtig es ist, die intrinsischen Fähigkeiten der verschiedenen Architekturen zu verstehen. Schließlich wollen wir durch die Schaffung eines standardisierten Bewertungsrahmens die Transparenz, Reproduzierbarkeit und Vergleichbarkeit innerhalb der MedMNIST+-Datensatzsammlung sowie die zukünftige Forschung verbessern.
Autoren: Sebastian Doerrich, Francesco Di Salvo, Julius Brockmann, Christian Ledig
[Preprint], [Publication], [Code], [Benchmark], [BibTeX](612.0 B)
F. Di Salvo, D. Tafler, S. Doerrich, C. Ledig, "Privacy-preserving datasets by capturing feature distributions with Conditional VAEs," BMVC 2024
Für die Weiterentwicklung von Deep-Learning-Anwendungen sind große und gut annotierte Datensätze unerlässlich. Diese sind jedoch oft kostspielig oder für eine einzelne Einrichtung unmöglich zu beschaffen. In vielen Bereichen, darunter auch im medizinischen Bereich, haben sich Ansätze durchgesetzt, die auf Datenaustausch basieren, um diese Herausforderungen zu bewältigen. Datenaustausch ist zwar wirksam, um die Größe und Vielfalt von Datensätzen zu erhöhen, er wirft jedoch auch erhebliche Datenschutzbedenken auf. Häufig verwendete Anonymisierungsmethoden, die auf dem k-Anonymitätsparadigma basieren, können die Datenvielfalt oft nicht bewahren, wodurch die Robustheit des Modells beeinträchtigt wird. In dieser Arbeit wird ein neuartiger Ansatz vorgestellt, der Conditional Variational Autoencoders (CVAEs) verwendet. Diese wurden auf Merkmalsvektoren trainiert, die aus großen, vorab trainierten Vision-Foundation-Modellen extrahiert wurden. Foundation-Modelle erkennen und repräsentieren effektiv komplexe Muster über verschiedene Domänen hinweg. Dadurch kann der CVAE den Einbettungsraum einer bestimmten Datenverteilung originalgetreu erfassen, um eine vielfältige, datenschutzkonforme und potenziell unbegrenzte Menge synthetischer Merkmalsvektoren zu generieren (zu samplen). Unsere Methode übertrifft traditionelle Ansätze sowohl im medizinischen als auch im natürlichen Bildbereich deutlich. Sie weist eine größere Datensatzvielfalt und eine höhere Robustheit gegenüber Störungen auf, während die Privatsphäre der Stichproben gewahrt bleibt. Diese Ergebnisse unterstreichen das Potenzial generativer Modelle, Deep-Learning-Anwendungen in datenarmen und datenschutzsensiblen Umgebungen erheblich zu verbessern.
Autoren: Francesco Di Salvo, David Tafler, Sebastian Doerrich, Christian Ledig
[Preprint], [Publication], [Code], [Bibtex](425.0 B)
F. Di Salvo, S. Doerrich, C. Ledig, "MedMNIST-C: Comprehensive benchmark and improved classifier robustness by simulating realistic image corruptions," ADSMI @ MICCAI 2025
Die Integration von Systemen auf Basis neuronaler Netze in die klinische Praxis ist aufgrund von Herausforderungen im Zusammenhang mit Domänengeneralisierung und Robustheit eingeschränkt. Die Computer-Vision-Community hat Benchmarks wie ImageNet-C als grundlegende Voraussetzung für die Messung der Fortschritte bei der Bewältigung dieser Herausforderungen etabliert. In der medizinischen Bildgebungsgemeinschaft fehlen ähnliche Datensätze weitgehend, da es an einem umfassenden Benchmark mangelt, der alle Bildgebungsmodalitäten und -anwendungen abdeckt. Um diese Lücke zu schließen, haben wir den Benchmark-Datensatz MedMNIST-C erstellt und als Open Source veröffentlicht. Er basiert auf der MedMNIST+-Sammlung und umfasst 12 Datensätze und 9 Bildgebungsmodalitäten. Mithilfe von simulierten, aufgaben- und modalitätsspezifischen Bildverfälschungen unterschiedlicher Schwere bewerten wir die Robustheit etablierter Algorithmen gegenüber realen Artefakten und Verteilungsverschiebungen umfassend. Darüber hinaus liefern wir quantitative Belege dafür, dass unsere leicht zu verwendenden künstlichen Verfälschungen eine leistungsfähige und effiziente Datenvergrößerung ermöglichen und somit die Robustheit des Modells verbessern. Im Gegensatz zu traditionellen, generischen Vergrößerungsstrategien nutzt unser Ansatz Domänenwissen und ist im Vergleich zu weit verbreiteten Methoden deutlich robuster. Mit der Einführung von MedMNIST-C und der Veröffentlichung der entsprechenden Bibliothek, die gezielte Datenvergrößerungen ermöglicht, tragen wir zur Entwicklung immer robusterer, auf die Herausforderungen der medizinischen Bildgebung zugeschnittener Methoden bei.
Autoren: Francesco Di Salvo, Sebastian Doerrich, Christian Ledig
[preprint], [code],[dataset], [bibtex](291.0 B)
S. Doerrich, F. Di Salvo, C. Ledig, "Self-supervised Vision Transformer are Scalable Generative Models for Domain Generalization", MICCAI, 2024
Trotz bemerkenswerter Fortschritte wurde die Integration von Deep Learning (DL)-Techniken in wirkungsvolle klinische Anwendungen, insbesondere im Bereich der digitalen Histopathologie, durch die Herausforderungen behindert, die mit dem Erreichen einer robusten Generalisierung über verschiedene Bildgebungsdomänen und -merkmale verbunden sind. Traditionelle Strategien in diesem Bereich wie Datenerweiterung und Fleckenfarbnormalisierung haben sich als unzureichend erwiesen, um diese Einschränkung zu beheben, was die Erforschung alternativer Methoden erforderlich machte. Zu diesem Zweck schlagen wir eine neuartige generative Methode zur Generalisierung von Histopathologiebildern vor. Unsere Methode verwendet einen generativen, selbstüberwachten Vision Transformer, um dynamisch Merkmale von Bildfeldern zu extrahieren und sie nahtlos in die Originalbilder einzufügen, wodurch neue, synthetische Bilder mit verschiedenen Attributen entstehen. Indem wir den Datensatz mit solchen synthetischen Bildern anreichern, wollen wir seine Ganzheitlichkeit erhöhen und eine verbesserte Generalisierung von DL-Modellen auf unbekannte Bereiche ermöglichen. Ausführliche Experimente mit zwei verschiedenen Histopathologiedatensätzen zeigen die Effektivität des von uns vorgeschlagenen Ansatzes, der den Stand der Technik deutlich übertrifft, und zwar beim Camelyon17-Wilds-Datensatz (+2%) und bei einem zweiten Epithelium-Stroma-Datensatz (+26%). Darüber hinaus heben wir die Fähigkeit unserer Methode hervor, mit zunehmend verfügbaren unbeschrifteten Datenproben und komplexeren, höher parametrischen Architekturen zu skalieren.
Autoren: Sebastian Doerrich, Francesco Di Salvo, Christian Ledig
[Preprint], [Publication], [Code], [BibTeX](612.0 B)
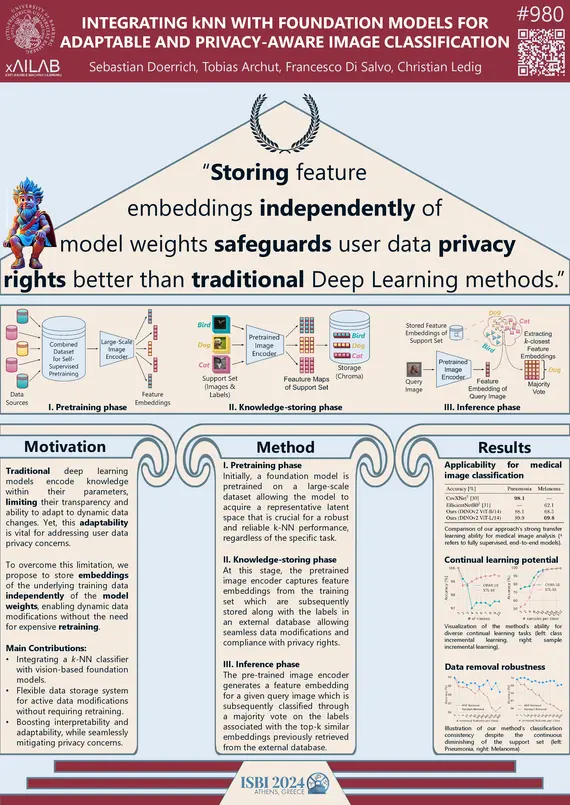
S. Doerrich, T. Archut, F. Di Salvo, C. Ledig, "Integrating kNN with Foundation Models for Adaptable and Privacy-Aware Image Classification", IEEE ISBI, 2024
Herkömmliche Deep-Learning-Modelle kodieren Wissen implizit, was ihre Transparenz und ihre Fähigkeit zur Anpassung an Datenänderungen einschränkt. Diese Anpassungsfähigkeit ist jedoch entscheidend für den Schutz der Daten der Nutzer. Wir beheben diese Einschränkung, indem wir die Einbettungen der zugrunde liegenden Trainingsdaten unabhängig von den Modellgewichten speichern und so dynamische Datenänderungen ohne erneutes Training ermöglichen. Unser Ansatz integriert insbesondere den k-Nearest-Neighbor-Klassifikator (k-NN) mit einem bildbasierten Foundation-modell, das selbstüberwacht auf natürlichen Bildern trainiert wurde, was die Interpretierbarkeit und Anpassungsfähigkeit verbessert. Wir stellen Open-Source-Implementierungen einer bisher unveröffentlichten Basismethode sowie unsere leistungssteigernden Beiträge zur Verfügung. Quantitative Experimente bestätigen die verbesserte Klassifikation in etablierten Benchmark-Datensätzen und die Anwendbarkeit der Methode auf verschiedene medizinische Bildklassifikationsaufgaben. Darüber hinaus bewerten wir die Robustheit der Methode in Szenarien mit kontinuierlichem Lernen und Datenentfernung. Der Ansatz ist sehr vielversprechend, um die Lücke zwischen der Leistung von Foundation-modellen und den Herausforderungen des Datenschutzes zu schließen.
Autoren: Sebastian Doerrich, Tobias Archut, Francesco Di Salvo, Christian Ledig
[Preprint], [Publication], [Code], [BibTeX](612.0 B)